Prima la scoperta che il DNA si avvolge su se stesso a guisa di doppia elica (1953). Poi il sequenziamento completo del genoma umano (2001). In mezzo, e successivamente, una marea di scoperte che di anno in anno hanno reso la genomica la disciplina del futuro, la leva principale delle promesse della Precision Medicine. Sarà mai possibile – si chiedono ora biologi, medici, informatici, farmacologi – penetrare nel minimo dettaglio della dimensione molecolare delle malattie? Smontarne e rimontarne il codice per cancellare quelle mutazioni, variazioni, impronte epigenetiche che accumulandosi segnano un destino di malattia?
Potremo, creando una mappa completa dei geni, e di come l’ambiente stesso agisce sulla tastiera dell’espressione genica, dei trascritti e delle proteine, mettere a punto farmaci personalizzati, che come chiavi molecolari possano aprire le serrature di ogni variante di tumore, di Alzheimer, di Parkinson, e di molti altri mali? Giusta o illusoria che sia, è questa l’ipotesi di lavoro della cosiddetta precision medicine, che negli Stati Uniti ha convinto il presidente Obama a lanciare nel dicembre 2015 la Precision Medicine Initiative, in Gran Bretagna circa un anno fa la One Hundred Thousand Genome e in Italia il progetto Human Technopole di IIT, che ora, dopo un lungo travaglio (ne abbiamo parlato in altri articoli), sta prendendo il via con la partecipazione di altri enti e università. Nello stesso periodo prende forma anche il progetto Genomi Italia ideato dalla biologa e senatrice Elena Cattaneo.
Actionable genoma
Per dare un volto a questo nuovo filone sentiamo Pier Giuseppe Pelicci, che per Human Technopole ha scritto la parte relativa alla genomica dei tumori. Sui geni del cancro Pelicci lavora da qualche decennio con risultati importanti, dalla famosa p66, che controlla la morte cellulare e che viene ribattezzata dai media il “gene della longevità”, alle importanti scoperte nel campo della comprensione e della cura della leucemia promielocitica acuta, la cui guarigione schizza dal 20 all’80% dei casi.
“Cos’è la genomica oggi?” mi chiede e si chiede dall’alto del suo metro e novanta circa, con la sua incancellabile “s” da umbro. “La genomica su cui oggi dobbiamo concentrarci è quella nota come ‘actionable genomics’, cioè quei geni su cui si può già agire con farmaci, che hanno cioè un significato clinico concreto”. Per il cancro i geni mutati su cui oggi si può agire attraverso un farmaco sono 161. La medicina in questione non è detto che sia risolutiva, ma in presenza di una mutazione in uno di questi geni risulta funzionare molto meglio. Poi vi sono – sempre secondo Pelicci – altri circa 200 geni che non sono vere e proprie mutazioni ma varianti alleliche che prevedono sensibilità a determinati farmaci. “Oggi siamo all’anno zero. Nel senso che noi curiamo i malati di cancro senza conoscere di fatto il loro assetto genico, quindi senza conoscere quei geni bersaglio di farmaci specifici. Certo per ora i pazienti che potrebbero avvantaggiarsi di queste conoscenze sono pochi, circa il 5% di tutti i malati di cancro. Ma la cosa incredibile è che solo il 5% di questo 5% viene curato con medicinali di nuova generazione efficaci ai loro geni mutati”. Dunque il primo obiettivo è di raggiungere tutti malati di tumore in Italia sottoponendoli a screening genomico alla ricerca dei loro “actionable genoma”, con l’obiettivo di dare il farmaco giusto alla persona giusta.
Ma c’è un secondo obiettivo: portare l’actionable genoma dal 5 a circa il 40%. Come? Passando in rassegna tutti i farmaci oncologici già esistenti, testandoli contro tumori fuori dalle loro attuali indicazioni terapeutiche ma compatibili perché nel malato si è trovata la stessa mutazione. “Esempio: esiste un farmaco efficace contro una mutazione responsabile del melanoma, il vemurafenib. Ma anche in determinati tumori del cervello si può trovare quella mutazione. Quindi, una volta ottenuto il profilo genetico dei malati si può fare uno screening virtuale su questi database e partire con nuovi trial clinici per riproporre il farmaco per una nuova indicazione terapeutica”. In questo modo, secondo Pelicci, si è relativamente certi di arrivare a curare in modo più efficace e preciso un terzo dei pazienti oncologici.
Terzo gradino, bello alto: lavorare sugli altri due terzi dei malati di cui ancora si ignorano le caratteristiche genetiche. “Partiamo col dire che le malattie sono molto eterogenee anche al loro interno, i tumori lo sono drammaticamente di più” spiega Pelicci. “E questa è anche la ragion d’essere, in prospettiva, di una medicina personalizzata. Colpire il bersaglio, e non a caso”. E se possibile agire prima della malattia. “Sono circa un centinaio i geni a noi noti che predispongono a malattia” continua Pelicci. “Per esempio, il BRCA1 ci dice che nel corso di una vita potrebbe svilupparsi nella persona con quella variante un cancro al seno con il 90% di probabilità”. Che fare? “Primo obiettivo: uno screening su scala nazionale dei familiari dei pazienti con queste mutazioni. Secondo obiettivo: espandere la ricerca dai familiari ad alcune popolazioni selezionate, per le quali è noto un nesso fra la condizione e la malattia oncologica, per esempio fra obesità e cancro alla mammella. “Mentre negli Stati uniti son partiti con un programma di screening a tappeto sulla popolazione, noi possiamo lavorare, almeno all’inizio su alcune categorie più rilevanti sul piano clinico”.
La ricerca di nuovi farmaci ritagliati sui nuovi geni predisponenti che man mano, anche grazie a una vasta collaborazione internazionale si andranno a identificare, è un processo lungo, lunghissimo per i malati, e costoso. Il progetto di Pelicci di Clinical genomics prevede anche un accesso precoce all’innovazione farmaceutica attraverso trial rapidi, anche da svolgere in parallelo su modelli animali personalizzati sull’assetto genomico del singolo paziente. Aspetto, questo, giudicato forse troppo ottimistico da parte di alcuni revisori internazionali del Progetto HT.
Immunità e cervello sotto la lente molecolare
Di grande rilevanza anche il programma di immunogenomica di HT, ideato da Alberto Mantovani (Humanitas University). Come è noto i processi infiammatori stanno alla base di tumori, malattie neurodegenerative altre malattie, una sorta di linguaggio comune che ha la sua chiave nel sistema immunitario e che il progetto si propone di scandagliare nelle sue diverse componenti omiche. In particolare, il programma prevede di “indagare la plasticità e la regolazione delle cellule immunitarie che interagiscono con l’ambiente del tumore (cioè dei linfociti tumore-infiltranti, i macrofagi, e di altre cellule mieloidi); di caratterizzare a livello di singola cellula i meccanismi molecolari e le reti sottostanti le risposte immunitarie innate e adattive al cancro; di definire dal punto di vista molecolare l’immunoediting del cancro, cioè le influenze reciproche fra cellule tumorali e cellule immunitarie. Infine, di elaborare nuovi modelli anche in vivo delle interazioni fra sistema immunitario e cellule tumorali, utilizzando anche organoidi tumorali ricostituiti con cellule immunitarie derivate dai pazienti”.
Per quanto riguarda la Neurogenomica, Stefano Gustincich, assieme ai colleghi neuroscienziati e neurologi delle istituzioni di ricerca e cura milanesi, propone un progetto di raccolta di campioni biologici, dati clinici e sequenze genomiche per poter classificare su basi molecolari i pazienti di malattie quali l’Alzheimer, il Parkinson e la Sclerosi Laterale Amiotrofica. Sono malattie altamente complesse, attualmente incurabili, che necessitano uno studio standardizzato su un alto numero di pazienti. Le cliniche neurologiche, inizialmente dell’area di Milano, coordinate dall’Istituto Neurologico Carlo Besta, sceglieranno i pazienti appropriati da includere nel progetto HT, li sottoporranno a esami clinici condivisi e alla raccolta di campioni biologici in modo standardizzato, formulando precise domande scientifiche. Collaboreranno attivamente con i Centri Computazionali e di Big data previsti nel progetto HT, al fine di creare un database ottimizzato per la medicina di precisione delle malatttie neurodegenerative. “Questo progetto servirà a creare una banca di tessuti, reagenti e dati genomici per la comunità scientifica e clinica nazionale. Per ogni paziente si procederà alla produzione di cellule staminali indotte che potranno essere distribuite ai ricercatori interessati: una risorsa estremamente preziosa in quanto permetterà di poter confrontare i dati sperimentali di laboratori diversi su campioni comuni conoscendo la sequenza genomica e le caratteristiche dei pazienti donatori” spiega Gustincich.
Progetto Genomi Italia
Attivato anch’esso a fine 2015 e oggi in via di definizione, il Progetto Genomi Italia si prospetta come un altro importante contributo alla precision medicine. È nato dalla constatazione che in Italia manca ciò che esiste già da tempo in molti paesi, ossia un progetto nazionale di sequenziamento genomico, che permetta di identificare meglio a livello sia di popolazione sia individuale quali siano gli eventuali biomarcatori genomici utilizzabili nella pratica clinica e in eventuali programmi di screening. Come dichiara il documento ufficiale di avvio del progetto “una base di conoscenza comune e specifica riguardante il nostro paese potrà contribuire a identificare varianti genetiche rare connesse a determinate malattie, alla risposta ai farmaci, al rischio e alla prevenzione di alcune patologie, allo sviluppo e all’esecuzione di test potenzialmente in grado sia di migliorare lo sviluppo di nuovi farmaci che di personalizzare la terapia”. Anche qui dunque il motivo conduttore è la medicina personalizzata, di cui il progetto dovrà favorire lo sviluppo realizzando un gigantesco archivio ragionato del DNA degli italiani. Non a caso tra i promotori ha avuto ed ha un ruolo chiave Elena Cattaneo, che con il suo gruppo di lavoro all’Università degli Studi di Milano ha conseguito risultati fondamentali per la comprensione della corea di Huntington, una malattia neurodegenerativa fatale, causata da una singola mutazione genica. Purtroppo per i malati di Huntington la strada verso la cura è ancora lunga, tuttavia gli studi di Cattaneo hanno chiarito aspetti chiave della funzione della huntingtina – una strana proteina che in forma mutata induce progressivamente la morte dei neuroni, mentre in forma non patologica svolge per queste cellule un fondamentale ruolo protettivo – contribuendo ad aprire nuove prospettive di terapia.
Oltre i geni
I geni non sono tutto. Man mano che lo si studia, il genoma appare come una realtà sempre più complessa, frutto dell’interazione di dimensioni fortemente integrate, di reti e relazioni che solo ora stiamo imparando a manipolare. Com’è noto, intimamente associati alla molecola di DNA si trovano quei blocchi di proteine (istoni) che regolano l’accensione dei geni. Attraverso diversi tipi di modificazione, l’ambiente circostante – la dieta, l’inquinamento, lo stress… – è in grado di variare l’espressione dei geni come un pianista su una tastiera. E’ questo, molto in soldoni, l’epigenoma. In molti casi le modifiche epigenetiche indotte dall’ambiente sono reversibili, in altri casi possono essere permanenti, trasmettendosi addirittura di generazione in generazione. “E non è detto che tali modificazioni siano tutte e solo negative, predisponendo a malattia” continua Pelicci. “Si possono anche ipotizzare interventi epigenetici migliorativi, preventivi. Si è osservato, per esempio, che un’alimentazione bilanciata ma povera, cioè basata su una restrizione calorica, aumenta – nel topo come nell’uomo, la longevità, o quanto meno può agire sulla crescita tumorale. Ebbene sia particolari regimi alimentari, sia alcuni farmaci ora allo studio che mimano la restrizione calorica, si presentano di fatto come interventi epigenetici. L’alimentazione è solo una delle possibili vie attraverso le quali l’ambiente può agire sull’espressione dei geni.
Il programma prevede quindi uno studio dell’epigenoma e dei meccanismi di interazione geni-ambiente.
Il programma di Pelicci prevede l’attivazione di screening epigenomici dei pazienti (cominciando dai malati che vengono curati negli ospedali milanesi che fanno rete con HT), una volta identificati compiutamente i biomarcatori epigenetici nel sangue. Anche il livello epigenomico, quindi, si presta sia a programmi di prevenzione (attraverso l’alimentazione, vedi oltre, l’esercizio fisico etc), sia di diagnosi precoce, sia di nuove terapie. “In realtà farmaci che agiscono sull’epigenoma esistono già. Per esempio molecole in grado di avere come bersaglio l’attività di enzimi che modificano la cromatina (istone demetilasi e deacetilasi)”.
La ricerca della divisione medica-oncologica di Human Technopole si rivolge quindi ai geni, all’epigenoma, così come al trascrittoma (frutto della trascrizione del DNA genomico sull’RNA, ne trasferisce il messaggio fuori dal nucleo della cellula rendendolo operativo) e alle proteine (proteoma) che ne sono il risultato finale. Tutti questi livelli vanno considerati insieme, nelle loro interazioni.
In Italia dal 2012 è attivo un importante progetto nazionale dedicato allo studio dell’epigenoma (Epigen: www.epigen.it), finanziato dal MIUR e coordinato da Giuseppe Macino. Il progetto raduna circa 70 gruppi di ricerca in tutta Italia, integrati in una rete in cui lavorano insieme CNR, università e fondazioni private, con piattaforme centralizzate per l’attività di sequenziamento e di supercalcolo. Fra gli obiettivi del progetto, studiare a livello di epigenoma malattie come progeria, sindrome di Williams, distrofia muscolare e diversi tipi di tumore; comprendere meglio le basi molecolari dei fenomeni di adattamento dei diversi organismi all’ambiente, di cui l’epigenetica è certamente la chiave (in fondo, la si potrebbe vedere come la rivincita di Lamarck su Darwin perché consente di assistere a mutamenti adattivi all’interno di una stessa generazione…). Infine Epigen lavora anche sulle possibili ricadute pratiche di queste conoscenze. “Il fatto che molti meccanismi epigenetici siano reversibili”, spiega Macino, “li fa diventare bersagli interessanti per nuove terapie, fra cui anche una nuova generazione di farmaci, i cosiddetti epidrug”.
Un google per la genomica
Più ci si addentra nello studio dei vari livelli dell’informazione genomica, maggiore diventa la sensazione di una quantità straordinaria di dati da dover dominare. Piergiuseppe Pelicci conferma: “La gestione dei dati rappresenta la vera sfida dell’era postgenomica, perché è la prima volta nella storia che ci troviamo di fronte a una massa tale di dati che stiamo imparando ora a integrare e interpretare, cercando di trarne da un lato interpretazioni cliniche, dall’altro una conoscenza personalizzata del paziente che metta insieme stili di vita, modifiche ambientali dell’epigenoma, mutazioni e differenze di alleli significative per la scelta del trattamento e delle scelte di prevenzione”.
Con i dati si misura quotidianamente Stefano Ceri, docente del Politecnico di Milano di computer science e grande esperto di genomic computing, tanto da da essere fra i pochissimi in Europa ad essersi aggiudicato un secondo grant dell’European Research Council (ERC) proprio su questo tema. Ceri lavora essenzialmente sulla messa a punto di una linguaggio informatico che consenta di superare le vecchie barriere dei programmi di bioinformatica e di consentire una lettura comune, universale, delle banche dati di genomi sparse per il mondo, da The Cancer Atlas del Broad Institute a Encode, a Roadmap Epigenomics. “Quello che stiamo facendo è integrare tutti questi dati e sottoporli a un’analisi terziaria, che ne faccia emergere regolarità, tratti distintivi, aspetti in qualche modo correlabili a malattie” spiega Ceri. Per questo il gruppo del Politecnico ha messo a punto un linguaggio – Il Genometric Query Language (GMQL) – che promette di essere il “google della genomica”, capace com’è di stabilire correlazioni fra diverse regioni del genoma. In una ricerca, per esempio, su 130.000 geni analizzati sono stati evidenziati 83 milioni di picchi di espressione che andranno poi vagliati individuando quelli potenzialmente correlati a tumori. GMQL è stato usato anche per setacciare i genomi della flora intestinale (il cosiddetto microbioma) di malati di fibrosi cistica, per comprendere meglio come una maggiore biodiversità microbica difenda dalla malattia.
Far parlare i dati può dare un impulso formidabile alla produttività e alla qualità delle ricerche biomediche, da qui il connubio sempre più stretto fra computer science e genomica. Che questo sia, potenzialmente, anche il business del futuro l’ha capito anche un big della computer science come Mike Stonebreaker – docente al MIT e Premio Turing 2015 – che a Cambridge ha fondato una start up, la Paradigm4, che setaccia dati biomolecolari per clienti come Novartis, Foundation Medicine e National Institutes of Health.
DNA 3D
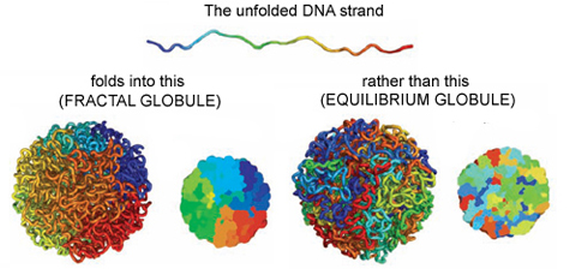
Oltre ai dati è la forma stessa del genoma a contenere informazioni fondamentali. Il gruppo di Ceri lavora anche su questo fronte, basandosi peraltro su scoperte recentissime, che non risalgono a prima di cinque anni fa. Come si comincia a dimostrare in alcuni tumori, il modo in cui il genoma si “piega” e forma dei loop ha un profondo significato biologico funzionale, maggiore ancora dei cromosomi. Spiega Ceri: “L’appallottolamento del genoma si conserva nelle specie ed è abbastanza caratteristico. E come alcune ricerche hanno mostrato nel 2012, ogni dominio topologico del genoma è legato a funzioni, e dai nostri studi sulle banche dati genetiche si osserva una correlazione fra espressioni dei geni e domini topologici di cui fanno parte”.
Sulla biologia computazionale lavora anche Andrea Cavalli, che per Human Technopole si è occupato del centro di Computational Life Sciences e dell’infrastruttura di Data Storage e High-Performance Computing. Dal centro computazionale, ci si aspetta la messa a punto di una visione complessiva e tridimensionale del genoma, dagli atomi alle basi, fino alla sua struttura complessa. Attraverso simulazioni al computer, l’intento è ricostruire la struttura tridimensionale del genoma e valutarne il suo comportamento nel tempo integrando la chimica e la fisica del fenomeno. I nuovi metodi multiscala che verranno sviluppati all’interno di questo centro permetteranno di studiare dal movimento degli elettroni fino ai riarrangiamenti strutturali della cromatina, con l’obiettivo ultimo di capire come la struttura tridimensionale del genoma possa essere correlata con la genesi e lo sviluppo delle malattie. L’ambiente virtuale permetterà di simulare l’interazione tra nuove piccole molecole e le strutture biologiche in esame, aprendo la strada alla scoperta di nuovi farmaci.
La chiave è la complessità
La medicina di precisione nasce dalla consapevolezza che non esiste un magic bullet, un gene o pochi geni responsabili della trasformazione delle cellule in cellula neoplastica. Su questo presupposto lavora da tempo il Centre for complexity and biosystems (CC&B) dell’Università di Milano, che studia tumori e molte altre patologie mettendo insieme biologia, fisica e informatica, con un approccio fortemente interdisciplinare.
“Per poter svolgere le proprie funzioni, la cellula utilizza una intricata e complessa rete di reazioni che comprende geni e proteine, a loro volta modulati da fattori esterni” spiegano la patologa Caterina La Porta e il fisico Stefano Zapperi, del CC&B. “Quello che anche noi, come altri gruppi nel mondo, stiamo cercando di fare è di decifrare questa rete, identificando i nodi più importanti (Hub) e la dipendenza del loro comportamento dall’ambiente circostante”.
La famacologia è cambiata in questi anni: ormai si parte da studi in silico, con il computer, e poi si passa al wet lab per confermare e chiarire meglio i risultati ottenuti. “Nel campo dei tumori, con questa strategia abbiamo ottenuto un risultato significativo in collaborazione con i colleghi della Cornell University, dimostrando come le cellule staminali tumorali siano in realtà uno stato cellulare modulato da un complesso network di fattori (microRNA in particolare)” spiega La Porta. “Agendo opportunamente su questi fattori è quindi possibile far cambiare stato alla cellula”.
Grazie a questo nuovo approccio che fa lavorare insieme biologia, fisica e computer science si è potuto anche dimostrare che le malattie neurodegenerative sono dovute ad una transizione di fase nel processo di aggregazione, regolato dall’ambiente. “Le malattie che stiamo studiando sono molteplici, ma in tutti i casi seguiamo rigorosamente un approccio quantitativo ai dati” concludono La Porta e Zapperi. “Questo significa che non ci accontentiamo solo di osservare un cambiamento, ma pretendiamo di misurarlo utilizzando tecniche di analisi statistica altamente sofisticate. Un aspetto su cui stiamo rivolgendo la nostra attenzione è l’interazione dei tumori con il sistema immunitario. Prevediamo che proprio da questa interazione possano emergere aspetti molto interessanti”.
Luca Carra – Agenzia Zadig